金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
基因组编辑技术发展概述
孙昌硕
华中农业大学生命科学技术学院 生物技术1302班 武汉 430070
摘要:自从上世纪中叶,沃森和克里克发现DNA双螺旋结构,人们就开始探索能够应用到生物体中的基因操作技术。最初的基因组编辑技术是利用寡核苷酸链和小分子,通过碱基配对来识别和切除靶标DNA,主要方法有化学法和内含子自剪接发。与此同时,人们发现了由锌指介导的DNA结合,它能引导FokI酶结合特定的序列同时发挥核酸酶的作用,从而切割靶标DNA。但由于锌指核酸酶 (ZFNs) 技术在使用过程中的不稳定性,人们于是ZFNs基于的切割原理创造了类转录激活因子效应物核酸酶(TALEN)技术,它的使用成本比ZFNs低,效率也更高。虽然TALEN技术有很多优点,但由于其使用的蛋白质向导在设计和合成上都比较困难,造成其推广受到限制。进入21世纪,美国科学家通过细菌和古菌的免疫防御系统,改进出来了一种利用RNA为向导的CRISPR -Case9系统基因组编辑技术,这一技术因其操作的简便性和使用的高效性,被全球的各个实验室广泛接受。近期,我国青年科学家韩春雨,独立研制出了以DNA为向导的NGAgo基因组编辑技术,受到各界的高度关注,甚至有人认为它将取代现今使用广泛的Case9系统。本文将对以上基因技术逐一进行介绍,并着重展示NgAgo这个新兴的基因组编辑技术,同时对它未来的发展进行展望。
关键词:基因组编辑技术 ZFNs TALEN CRISPR-Case9 NgAgo
正文
基因组编辑技术的真正形成距今不到五十年的时间,但这一领域已经衍生出了至少三代基因组编辑技术,并且今年五月报道的NgAgo基因组编辑技术有望成为第四代基因组编辑技术,这距离第三代基因组编辑技术——CRISPR-Case9系统的形成仅仅只有4年的时间。由此可见科学家们对基因组编辑技术的热情,以及这一领域高速发展不断适应研究和生产需求的现象。回顾基因组编辑技术的发展历史可以追溯到1951年,沃森和克里克发现了DNA双螺旋结构,这一伟大的发现激发了科学家们第DNA进行人工改造的想法;1957年,Rich和他的同事描述了三螺旋结构的形成(1),这为后面利用碱基配对的原理,通过寡核苷酸链和小分子识别待切割的靶基因序列提供了理论依据。1979年,Scherer 和Davis通过同源重组的方法,利用质粒将外源基成功转化到酿酒酵母的基因组中,并且外源基因能够正常的表达(2),这标志着基因组编辑技术的正式形成。就在同源重组基因组编辑技术兴起的同时,锌指核酸酶 (ZFNs) 技术也在逐渐形成。1996年,Kim等人将锌指蛋白同一种限制性内切酶Fok I的切割结构域融合后,创造出一种能利用锌指结构够识别特异DNA序列,并将其切割的基因组编辑技术(3)。由于锌指核酸酶的构建和筛选过程复杂,并且使用过程中错配的可能性大等原因,使得锌指技术使用受到限制。但这没有减弱科学家们对基因组编辑技术的热情,相反促使他们基于锌指核酸酶 技术,2009年,发展出了类转录激活因子效应物核酸酶(TALEN)技术(4-6),TALEN相比ZFN最大的不同是它使用氨基酸序列作为向导识别靶基因序列,相比用锌指结合靶基因,TALEN的特异性更高,使用成本更低。TALEN技术出现后得到了广泛的使用,但好景不长,一种操作更加方便,稳定性更高等更加优秀的基因组编辑技术在其兴起后不到两年的时间就将其主导地位取代,这就是2012年,M. Jinek等人研制出的CRISPR-Case9系统,一种用RNA作为向导的DNA限制性内切酶(7)。正当科学界在为CRISPR-Case9系统的产生而欢欣鼓舞和不断谋求将其完善的同时。中国青年科学家韩春雨,于今年五月发表了关于格氏嗜盐碱杆菌AGO蛋白通过DNA介导的基因组编辑技术,即现在轰动全球学术界的NgAgo基因组编辑技术(8),它相比Case9具有DNA序列介导,稳定性更好,可以在人体温的条件下操作,相比Case9更适用于在哺乳动物中进行基因编辑等许多优点,由于刚刚兴起,其很多方便不够完善,但相信其未来会迅速发展成为一项主流基因组编辑技术。基因组编辑技术的发展史可谓惊心动魄,各种新技术接踵而至,一些技术还未来得及完善很快就被另一种新技术取代,这体现了科学家们不断谋求更优秀的基因组编辑技术的热情,同时也说明了科学研究和生产活动对这一技术的热切需求。所以,基因组编辑技术将会在很长的一段时间内成为学术热点,更新速度可能超出想象,它们可能承接现有的技术,变得更加完善,也可能从一些生物中发现并改造成可供使用的新技术,但不论怎样,它们共同的目标应该都是使基因编辑更加稳定、高效、廉价,使转基因技术更加安全,能够更好的服务人们的生活。
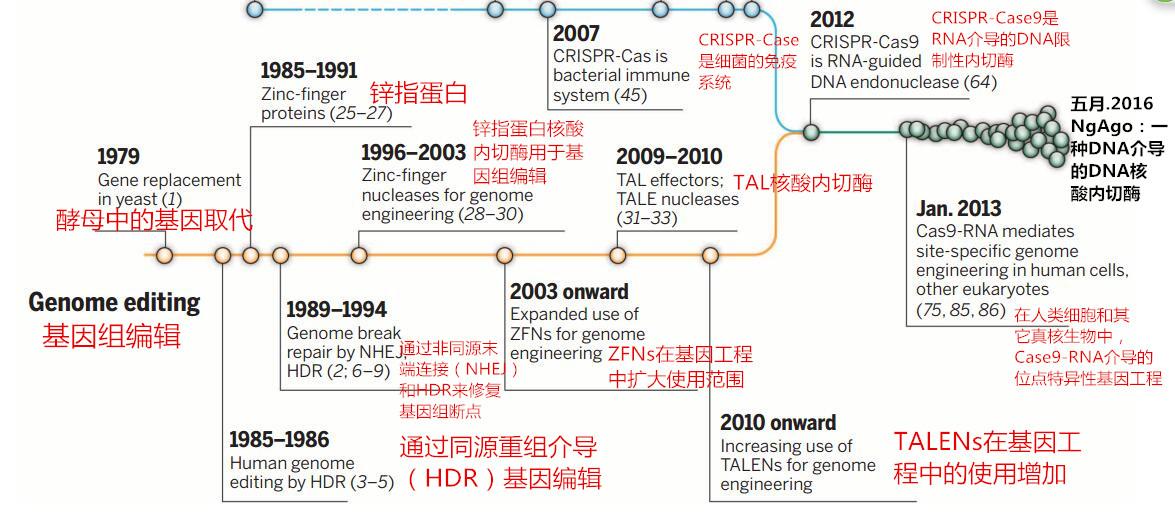
图1,基因组编辑技术的发展历程。(图片来源:Science. 2014 Nov 28;346(6213):1258096. doi: 10.1126/science.1258096.Figur1)
最早的基因组编辑技术——化学法和同源重组发
化学法主要有两种。第一种是将寡核苷酸链与能够剪切核酸的化学物质偶联或者是与争光霉素和补骨脂素交错连接,再用这些小链去结合靶基因,实验表明它们能在酵母和哺乳动物细胞中进行位点特异性的染色体修饰。(9)另一种化学方法是用能识别特定位点的核酸短肽和聚酰胺偶联切割反应剂,如争光霉素,它们结合到基因组后能够对结合的位点进行修饰。(10-12)同源重组法是基于碱基配对和内含子自剪切的方法来在DNA水平和RNA水平改造序列。虽然这些技术的出现并没有得到广泛关注和使用,但它们阐明了基于碱基配对进行的位点特异性基因编辑的可靠性,为之后的基因组编辑技术的开发奠定了基础。
第一代人工核酸内切酶——锌指核酸酶 (ZFNs) 技术
随着科学的发展,科学家们发现一种在许多蛋白质中广泛存在的蛋白质基序(Motif),它们能够形成介导蛋白质与核酸相互作用的特殊结构,这就是锌指(ZF),每个锌指可识别三个连续的核苷酸。通过将这些能形成ZF的氨基酸序列的编码序列串联起来,所形成的蛋白能够特异性识别并结合DNA,这种蛋白就是锌指蛋白(ZFP)。锌指蛋白只具有向导作用,没有切割作用。因此,科学家们将一种限制性内切酶Fok I的编码序列连接到锌指蛋白编码序列的3´端,得到一种锌指蛋白C端与Fok I相连的人工融合蛋白。一般一个锌指核酸酶(ZFNs)具有三个锌指结构和一个Fok I的切割功能结构域,作用位点的DNA序列长度在5~7bp,根据作用位点两侧的相邻链序列,筛选能与之正链和负链各一条特异性结合的两种ZFNs,当这两种ZFNs与DNA结合后,其C端的Fok I切割结构域会在作用位点形成二聚体,并发挥功能,使双链断裂,这会造成非同源末端连接(NHEJ)和同源重组(HR),于是作用位点的基因发生改变,完成基因编辑。
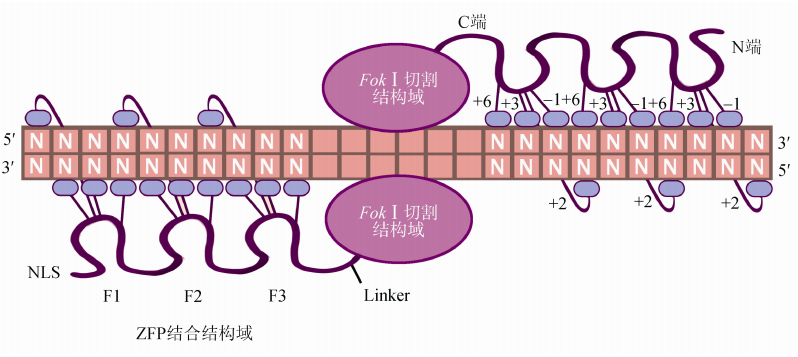
(图片来源:肖安. 人工锌指核酸酶介导的基因组定点修饰技术. 遗传, 2011,33(7): 第667页.)
图2,锌指蛋白发挥功能示意图。两个锌指核酸酶分别结合作用位点的两侧序列,其中一个结合正链,另一个结合负链,其N端与DNA链的3´端同侧,C端的Fok I切割结构域在作用位点耦合形成二级结构发挥功能。
第二代人工核酸内切酶——类转录激活因子效应物核酸酶(TALEN)技术
ZFNs技术是一种有效的基因组编辑技术,目前已经在小鼠、斑马鱼、拟南芥等许多生物中试验成功,并能产生稳定遗传的突变。但因为其针对特定序列的锌指蛋白(ZFP)的设计和筛选工作量大,且锌指蛋白容易发生错配等原因,造成其不能够被广泛接受。这促使科学家们寻找一种能更方便设计,与DNA序列结合特异性更强的蛋白质基序(motif)。最终,类转录激活因子效应物(TALE)应运而生,这是一种从植物病原体——黄单胞杆菌(Xanthomonas spp.)中分离出来的蛋白(18,19),他们发现这种蛋白能够通过特异性结合宿主DNA,调控其表达来提高病原体的致病性。TALE蛋白与锌指蛋白最大的不同体现在,它的编码序列中部包含一段很长的串联排列的重复序列,这段DNA序列编码的氨基酸能特异性识别并结合到作用位点上,并且这是利用氨基酸残基来识别核酸,每两种氨基酸残基对应一种类型的核苷酸(20)。这相比使用锌指结合DNA序列更加稳定,特异性也更高。
TALE蛋白的编码序列除了中部的串联重复序列外,其5´端具有核定位信号(NLS),3´端具有转录激活结构域(AD),研究人员将其3´的AD序列换成编码Fok I切割结构域的DNA序列,这ZNFs有异曲同工之处。经过改造后编码序列经过转录翻译后得到一个TALE蛋白与Fok I切割结构域融合的人工核酸内切酶,也就是我们现在熟知的类转录激活因子效应物核酸酶(TALEN)。TALEN的工作原理与ZFNs相似,也是TALE先结合到作用位点两侧的正或负链DNA上,然后Fok I蛋白在作用位点形成二聚体发挥切割功能,之后损伤的作用位点DNA激活HR或NHEJ,使作用位点的基因序列发生改变。
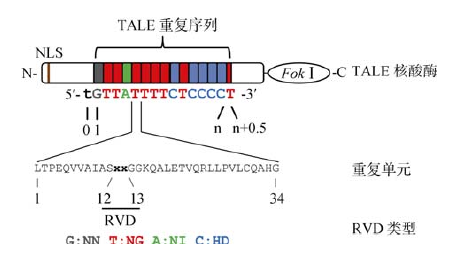
图片来源:沈延, 肖安, 黄鹏等. 类转录激活因子效应物核酸酶_TALEN_介导的基因组定点修饰技术. 遗传, 2013,35(4): 第397页
图3,TALEN蛋白的结构示意图。从左至右,N端为核定位信号(NLS);中部为串联重复的氨基酸序列,能够特异性识别氨基酸;C端为Fok I蛋白,为非特异性切割的限制性核酸酶,当两个Fok I蛋白聚在一起时发挥功能。
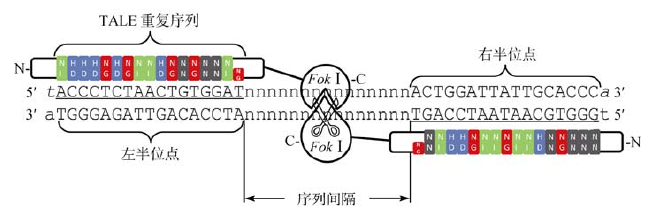
图片来源:沈延, 肖安, 黄鹏等. 类转录激活因子效应物核酸酶_TALEN_介导的基因组定点修饰技术. 遗传, 2013,35(4): 第398页
图4,TALEN发挥功能示意图。TALE重复序列与作用位点两侧序列特异性结合,分别结合在正负链上,N端指向DNA的5´端,C端两个Fok I在作用位点形成二聚体,并发挥作用。
第三代人工核酸内切酶——CRISPR-Cas9系统
如果要问21世纪初最伟大的发现有哪些?那么CRISPR-Cas9系统当之无愧是其中之一。CRISPR-Cas9系统是一种利用RNA介导的基因组编辑技术,相比ZFNs和TANEN需要大量的蛋白质工程实验来产生符合靶基因作用位点的氨基酸序列, CRISPR-Cas9系统只需要改变RNA的序列即可,相比前两者容易得多,并且通过碱基配对使得特异性更高,向导序列与作用位点结合得更加稳定,更重要的是它能在活体生物中完成基因组编辑。或许是因为具有许多的优点,虽然CRISPR-Cas9系统比TALEN晚出现三年,但它在很短的时间内取代TALEN成为实验室中的主要基因组编辑技术,并在不断的完善和发展,目前已经在人类和其它哺乳动物细胞中成功进行了位点特异性的DNA切割,未来有望解决一些人类相关疾病。同时在商业上,CRISPR技术已经多种转基因农作物中,利用它的可以将转基因作物的相关外源基因,在发挥作用后敲除,这样在收获的农产品中检测不到转入的外源基因,这有望使转基因食品更加安全,能够被更广泛的接受。
CRISPR全称是clustered regularly interspaced palindromic repeat,中文意思是成簇管理的有间隔回文重复序列,这样称呼它是因为它的编码基因中具有许多病毒基因相关的序列。细菌在进化的过程中,发展出了吸收病毒基因片段,并在相关病毒再次入侵时,转录相应序列,利用其引导Case蛋白与病毒基因结合并将其切割,影响其表达,从而达到免疫防御的作用(24)。CRISPR-Cas9技术就是从细菌抵抗的病毒和外源DNA入侵的免疫防御系统发展而来的,在细菌中一共有三种类型的CRISPR-Cas系统,研究者进过比较,发现II型系统最适合用于基因组编辑,经过改造后,得到了现在的CRISPR-Cas9系统(22-23)。CRISPR-Cas9系统由Case9酶和向导RNA两部分组成。Case9酶能够通过其内部的两个催化位点使双链DNA断裂,所切割序列靠近靶基因上的邻近预先间隔区域(PAM),它能与向导RNA(sgRNA)匹配,长度在20bp;向导RNA由两种RNA组成,其中一部分是来自于CRISPR自身基因转录出来的RNA(crRNA),它负责识并结合作用位点DNA序列,另一部分是从其它DNA中另行转录出来的RNA(tracrRNA),能够结合crRNA的部分序列和Case9蛋白,起稳定CRISPR-Cas9的作用。
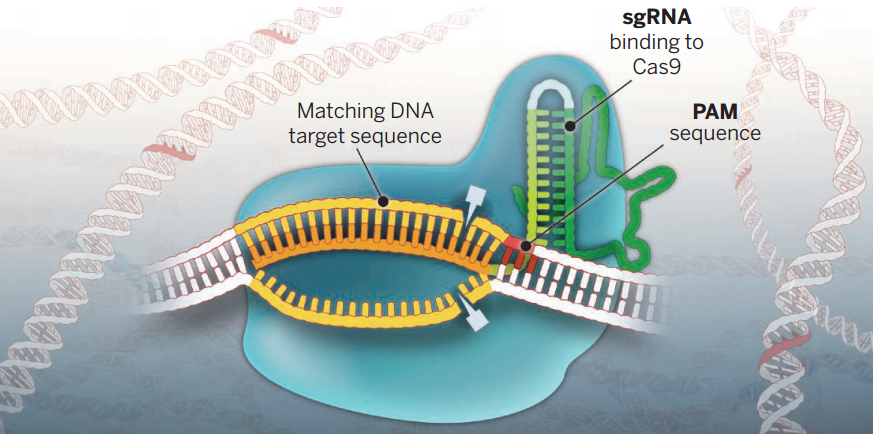
图片来源:Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9.Science. 2014 Nov 28;346(6213):1258096. doi: 10.1126: pmid:25430774
图5.Case9蛋白结构示意图。从图中可以看到一个完整的Case9蛋白包括Case9酶(蓝色),crRNA (亮绿色,其中与DNA结合的部分显示为橙色),tracrRNA (暗绿色);作用位点中可以看到,与crRNA结合的DNA序列(金色)是被修饰的部分,PAM序列(红色)影响Case酶的对作用位点DNA序列的切割。
明日之星——NgAgo-gDNA基因编辑技术
就在研究者们沉浸在CRISPR-Case9系统带来的便利,同时谋求不断完善它的时候。我国青年科学家韩春雨不甘现状,在今年五月,报道了一项可以与之比肩甚至更优越的基因编辑技术,轰动了全球学术界。这就是如今受到学术界高度关注的新兴基因组编辑技术NgAgo-gDNA基因编辑技术。
NgAgo(Natronobacterium gregoryi Argonaute) 是一种来源于格氏嗜盐碱杆菌(Natronobacterium gregoryi)的能够切割DNA靶标的Ago蛋白,简称NgAgo.Ago。这种蛋白是核酸内切酶中的一个家族,通过结合5´磷酸化的短的线性单链核苷酸(ssDNA/sgRNA)作为向导来切割靶标(27)。虽然,大多数Ago蛋白与sgRNA相关联,并在RNA沉默中发挥重要作用,但存在一些Ago蛋白能够结合ssDNA,发挥切割靶标DNA的功能(25-26)。韩春雨实验室根据文献报道,了解到来源于嗜热细菌(Thermus thermophilus)和火球菌(Pyrococcus furiosus)的Ago蛋白:TgAgo和PfAgo,二者中任意一个与5´磷酸化的ssDNA结合后,都能在试管中特异的切割靶基因(25-26)。但它们二者发挥功能都需要较高的反应温度(>65℃),这妨碍了它们在哺乳动物细胞中发挥作用。为了找到适用于编辑哺乳动物基因组的Ago蛋白,他们通过美国国家生物技术中心(NCBI)的位置特异性迭代查询局部序列排比检索工具(PSI-Blast),使用TtAgo和PfAgo的氨基酸序列在非冗余蛋白序列数据库中鉴定到一个从噬盐噬碱古细菌N. gregoryi SP2中分离出来的Ago蛋白(NgAgo,蛋白质注册号AFZ73749.1)他们以NgAgo为候选蛋白,进行功能验证,确定NgAgo是一种由DNA引导的,能够在37℃进行位点特异性切割的核酸内切酶(8)。
NgAgo之所以受到高度关注主要原因是因为它在很多方面优于现在流行的CRISPR-Case9系统。主要体现在以下三点:1.NgAgo与Case9都在抑制基因表达和防御外源DNA入侵的过程中发挥重要的作用(28),但Case9仅存在于原核生物中,而Ago蛋白质家族在生物进化过程中得到保留,存在于几乎所用的生物中,这使得NgAgo更易于在真核生物中使用;2.NgAgo与向导DNA(gDNA)结合不需要gDNA形成特殊的结构,而Case9结合的向导RNA(gRNA)需要在3´端形成RNA-RNA杂交结构,用来确保Case9能正确结合;最后,设计和合成用于引导NgAgo的gDNA序列很容易,但对于Case9需要的gRNA序列来说却很难。3.通过质粒产生相同数量的gDNA和gRNA,gDNA需要的质粒数量仅为gRNA需要的质粒数量的1/10(8)。综合以上三点,可以发现NgAgo相比Case9使用更加方便,想到序列的设计和合成也更加容易。
韩春雨实验室通过实验证明,NgAgo除了操作更加方便以外,其脱靶率更低,具体体现在一下两点:1.对向导DNA和靶标DNA间的错配低耐受。gDAN上的任意一个核苷酸与靶标DNA的核苷酸不匹配,都会严重减弱NgAgo对靶标的切割效率造成。尤其是存在三个及以上的错配位点时,将完全阻碍NgAgo发挥切割功能。此外,或许是因为gDNA链上的8号位核苷酸可能是NgAgo起始切割靶标DNA的位置,所以,当8号位核苷酸与靶标错配时,靶标DNA将不能够被切割;2.NgAgo蛋白只忠实于它在表达的过程中所结合的gDNA。gDNA只能在NgAgo蛋白质表达的过程中与其结合,并且,一旦二者结合,NgAgo-gDNA在37 ℃的情况下就不会再更换gDNA。但在55 ℃的情况下,NgAgo-gDNA复合体能与其它gDNA 一起孵育一小时后,会发生gDNA交换。然而实验发现,发生gDNA交换的NgAgo-gDNA复合体切割核苷酸序列的能力会严重降低,这或许是因为发生gDNA交换过程需要的温度会导致NgAgo蛋白质变性,甚至当55℃孵育时间过长时,会导致NgAgo的核酸酶活性丢失。这些机制降低了向导DNA发生错配或者被替换造成错误切割的可能(8)。
最重要的是,NgAgo在应用到哺乳动物基因组编辑中具有巨大的优势:首先,NgAgo能在37 ℃发挥功能。这一温度和大多数哺乳动物的正常体温相近。另外,哺乳动物细胞中几乎不产生能与NgAgo结合的5`磷酸化ssDNA。因为NgAgo发挥切割靶基因的功能需要5`磷酸化的短单链DNA(ssDNA)介导(最佳长度为24bp), 而5`磷酸化的ssDNA在哺乳动物细胞中几乎没有,这使得细胞中的同源核苷酸错误介导NgAgo切割细胞中同源序列的可能性被降到最低(8)。
目前,NgAgo技术刚刚兴起,很多方面还不够完善,但就目前观察到有点来说,它很有希望超越现今主流的基因编辑技术CRISPR-Case9系统成为第四代基因组编辑技术,我们将拭目以待。另外值得注意的是,韩春雨实验室是一个实验经费严重缺乏、实验室人手不足和实验设备相对落后的普通大学实验室,但他凭借自己不懈努力和不惧权威的气魄,最终钻研出这样轰动世界的能与世界顶尖实验比肩的实验结果是非常了不起的,这种朴素的专研精神值得每个生物科研工作者认真学习。
展望
基因组编辑技术在不到五十年的时间内经历了至少三代技术的变更,未来还将有更多更高效的基因编辑技术产生。如今的研究者主要是从自然界的生物(细菌,真菌)中寻找可用于设计基因组编辑系统的相关蛋白,依据其原理进行人工改造其部分结构后得到人工核酸内切酶,未来研究者们可能根据先前的经验,自行创造自然界中不存在的高效人工核酸内切酶。
现在,科学家们追求的是适用性更广的人工核酸内切酶,将来可能会根据特殊的需求,制作专一性很强的基因组编辑工具。不管基因组将来发展出怎样的新技术,发生多大的变化。可以肯定的是,未来五十年甚至更久远,基因组编辑技术依然会是科学界研究的热点,因为它不仅是一种生物学的研究工具,还是用来进行农业生产和医药卫生的重要手段,无论在作物改良、畜牧育种,还是在遗传疾病防治、病毒疫苗生产以及许多相关领域中都将发挥巨大的重用,可以说它将是人类今后生产生活必不可少的一门工具,但其使用带来的技术安全性和相关的伦理问题始终需要研究者进行关注,并努力解决。
致谢
感谢林拥军老师传授我基因工程的相关知识,让我更好的了解这一技术的能容和作用,并通过这次作业督促我对基因组编辑技术有了更全面深入的了解,使我对这一领域产生了更感兴趣,相信基因组编辑技术将会对我今后的学习生活和日常生活产生重要影响。同时感谢本班的李桐同学为我提供了TALEN技术的相关文献资料,让我更好的认识了ZFNs和TALEN这两种基因组编辑技术。
参考文献:
1.G. Felsenfeld, D. R. Davies, A. Rich, Formation of a threestranded polynucleotide molecule. J. Am. Chem. Soc. 79, 2023–2024 (1957). doi: 10.1021/ja01565a074
2.S. Scherer, R. W. Davis, Replacement of chromosome segments with altered DNA sequences constructed in vitro. Proc. Natl. Acad. Sci. U.S.A. 76, 4951–4955 (1979). doi: 10.1073/pnas.76.10.4951; pmid: 388424
3. Y. G. Kim, J. Cha, S. Chandrasegaran, Hybrid restriction enzymes: Zinc finger fusions to Fok I cleavage domain. Proc. Natl. Acad. Sci. U.S.A. 93, 1156–1160 (1996). doi: 10.1073/ pnas.93.3.1156; pmid: 8577732
4.J. Boch et al., Breaking the code of DNA binding specificity of TAL-type III effectors. Science 326, 1509–1512 (2009). doi: 10.1126/science.1178811; pmid: 19933107
5. M. J. Moscou, A. J. Bogdanove, A simple cipher governs DNA recognition by TAL effectors. Science 326, 1501 (2009). doi: 10.1126/science.1178817; pmid: 19933106
6. M. Christian et al., Targeting DNA double-strand breaks with TAL effector nucleases. Genetics 186, 757–761 (2010). doi: 10.1534/genetics.110.120717; pmid: 20660643
7. M. Jinek et al., A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816–821 (2012). doi: 10.1126/science.1225829; pmid: 22745249
8. Feng Gao, Xiao Z Shen, Feng Jiang, Yongqiang Wu & Chunyu Han. DNA-guided genome editing using the Natronobacterium. Nat Biotechnol. 2016 May 2. doi: 10.1038/nbt.3547.
9. S. A. Strobel, P. B. Dervan, Site-specific cleavage of a yeast chromosome by oligonucleotide-directed triple-helix formation. Science 249, 73–75 (1990). doi: 10.1126/ science.2195655; pmid: 2195655
10.J. Cho, M. E. Parks, P. B. Dervan, Cyclic polyamides for recognition in the minor groove of DNA. Proc. Natl. Acad. Sci. U.S.A. 92, 10389–10392 (1995). doi: 10.1073/ pnas.92.22.10389; pmid: 7479790
11. A. F. Faruqi, M. Egholm, P. M. Glazer, Peptide nucleic acidtargeted mutagenesis of a chromosomal gene in mouse cells. Proc. Natl. Acad. Sci. U.S.A. 95, 1398–1403 (1998). doi: 10.1073/pnas.95.4.1398; pmid: 9465026
12. J. M. Gottesfeld, L. Neely, J. W. Trauger, E. E. Baird, P. B. Dervan, Regulation of gene expression by small molecules. Nature 387, 202–205 (1997). doi: 10.1038/387202a0; pmid: 9144294
13.J. Yang, S. Zimmerly, P. S. Perlman, A. M. Lambowitz, Efficient integration of an intron RNA into double-stranded DNA by reverse splicing. Nature 381, 332–335 (1996). doi: 10.1038/381332a0; pmid: 8692273
14. S. Zimmerly et al., A group II intron RNA is a catalytic component of a DNA endonuclease involved in intron,mobility. Cell 83, 529–538 (1995). doi: 10.1016/0092-8674 (95)90092-6; pmid: 7585955
15. B. A. Sullenger, T. R. Cech, Ribozyme-mediated repair of defective mRNA by targeted, trans-splicing. Nature 371, 619–622 (1994). doi: 10.1038/371619a0; pmid: 7935797
16.Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering
with CRISPR-Cas9.Science. 2014 Nov 28;346(6213):1258096. doi: 10.1126: pmid:25430774
17.肖安,胡莹莹, 王唯晔等. 人工锌指核酸酶介导的基因组定点修饰技术. 遗传, 2011,33(7): 665-683.
18.Bibikova M, Golic M, Golic KG, Carroll D. Targetedchromosomal cleavage and mutagenesis in Drosophilausing zinc-finger nucleases. Genetics, 2002, 161(3): 1169–1175.
19. Rouet P, Smih F, Jasin M. Expression of a site-specificendonuclease stimulates homologous recombination inmammalian cells. Proc Natl Acad Sci USA, 1994, 91(13):6064–6068.
20.Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, KayS, Lahaye T, Nickstadt A, Bonas U. Breaking the code ofDNA binding specificity of TAL-type III effectors. Science, 2009, 326(5959): 1509–1512.
21.沈延, 肖安, 黄鹏等. 类转录激活因子效应物核酸酶_TALEN_介导的基因组定点修饰技术. 遗传, 2013,35(4): 295-309.
22.M. Jinek et al., A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816–821 (2012). doi: 10.1126/science.1225829; pmid: 22745249
23. G. Gasiunas, R. Barrangou, P. Horvath, V. Siksnys, Cas9- crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. U.S.A. 109, E2579–E2586 (2012). doi: 10.1073/ pnas.1208507109; pmid: 22949671
24.Y. Ishino, H. Shinagawa, K. Makino, M. Amemura, A. Nakata, Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J. Bacteriol. 169, 5429–5433 (1987). pmid: 3316184
25. Swarts, D.C. et al. DNA-guided DNA interference by a prokaryotic Argonaute. Nature 507, 258–261 (2014).
26. Swarts, D.C. et al. Argonaute of the archaeon Pyrococcus furiosus is a DNA-guided nuclease that targets cognate DNA. Nucleic Acids Res. 43, 5120–5129 (2015).
27. Swarts, D.C. et al. The evolutionary journey of Argonaute proteins. Nat. Struct. Mol. Biol. 21, 743–753 (2014).
28. Makarova, K.S., Wolf, Y.I., van der Oost, J. & Koonin, E.V. Prokaryotic homologs of Argonaute proteins are predicted to function as key components of a novel system of defense against mobile genetic elements. Biol. Direct 4, 29 (2009).
原文地址:https://zhuanlan.zhihu.com/p/21321196 |
|
 /3
/3 